编辑 | 萝卜皮
准确预测蛋白质和配体之间的结合亲和力对于药物发现至关重要。图神经网络(GNN)的最新进展在学习蛋白质-配体复合物的表示以估计结合亲和力方面取得了重大进展。为了提高 GNN 的性能,经常需要从几何角度研究蛋白质-配体复合物。
虽然「现成的」GNN 可以包含分子的一些基本几何结构,例如距离和角度,通过将复合体建模为同亲图,这些解决方案很少考虑更高级别的几何属性,例如曲率和同源性,以及异亲相互作用。
为了解决这些限制,之江实验室、百度大数据以及香港科技大学的研究人员引入了基于曲率的自适应图神经网络(CurvAGN)。该 GNN 包含两个组件:曲率块和自适应注意力引导神经块(AGN)。
曲率块对多尺度曲率信息进行编码,然后AGN基于自适应图注意力机制,将包括角度、距离和多尺度曲率、长程分子相互作用和图异质性在内的几何结构纳入蛋白质-配体复合体表示中。
该研究以「CurvAGN: Curvature-based Adaptive Graph Neural Networks for Predicting Protein-Ligand Binding Affinity」为题,于 2023 年 10 月 5 日发布在《BMC Bioinformatics》。

蛋白质-配体结合亲和力预测存在挑战
蛋白质-配体结合亲和力预测是药物发现的关键步骤。它使研究人员能够在进行昂贵且耗时的实验之前识别潜在的候选药物并优化其特性。三维 (3D) 结构蛋白数据的可用性不断增加,为基于结构的药物发现提供了新的范式,并且 3D 结构信息已被证明可以促进药物设计。
人们已经开发了各种计算方法来从蛋白质-配体复合物中学习 3D 结构信息。这些方法的范围从分子对接到更复杂的机器学习和深度学习方法。对接方法已被广泛采用,具有用于结合亲和力预测的评分功能,但其准确性也限制了对接方法的潜在应用。传统的机器学习算法与手工制作的特征有时可以提供不错的性能,但由于大量特征工程的成本,它们很难扩展。
为了对 3D 空间结构进行建模,许多深度学习方法将复杂的数据划分为 3D 网格数据,并应用 3D 卷积神经网络(3D CNN)来提取有用的特征。这些方法在预测结合亲和力方面表现出比传统的基于机器学习的模型更好的性能。然而,当使用 3D 矩形网格表示时,复合体中原子的稀疏分布可能会导致计算效率低下。
将蛋白质-配体复合物建模为节点对应于原子的图是一种自然而有效的方法。图神经网络(GNN)在表达图结构方面表现出了卓越的能力,研究人员在整合空间信息以增强其表达能力方面做出了相当大的努力。空间图卷积网络利用 3D 坐标对复合体的结构进行建模。
然而,基于坐标的模型的输出可能会受到坐标旋转的负面影响。这个限制可以通过距离感知 GNN 来解决,它只考虑距离。但这些模型可能不足以准确模拟 3D 结构以进行结合亲和力预测。基于定向消息传递的 GNN 已被提出来解决这一限制。
这些模型结合了角度和距离信息,这已被证明对于分子的经验势至关重要。虽然这些模型提供了改进的预测性能,但它们的准确性还有很大的潜力进一步提高。
由于蛋白质-配体结合亲和力是由其绝对结合自由能决定的,而绝对结合自由能主要由曲率指定,因此有必要将曲率信息纳入图形表示中以提高预测准确性。曲率的概念与流形的几何形状密切相关,并且已经做出了一些努力来推广图的曲率。
基于这种概括,科学家提出了两种不同的基于曲率的图神经网络,并且它们在基线数据集上表现良好。生物分子通常表现出层次和多尺度结构,这需要多尺度表示来准确表征它们的相互作用。这意味着图的多尺度曲率更合适。然而,将多尺度曲率纳入 GNN 来预测结合亲和力仍然是一个悬而未决的研究问题。
此外,许多研究已经认识到蛋白质-配体复合图的异质性,并努力将这种异质性纳入其图神经网络中。但是,人们经常忽视该图并不是严格同质的,因为相邻节点可能不相似。基于同质假设的图神经网络无法有效地学习异质性,异质性是链接节点具有不同特征的属性。因此,先前关于结合亲和力的研究未能捕获异质性。
基于曲率的自适应图神经网络应对挑战
为了解决上述挑战,之江实验室、百度大数据以及香港科技大学的合作团队提出了一种新颖的基于曲率的自适应图神经网络(CurvAGN)来预测蛋白质-配体结合亲和力。CurvAGN 包括曲率块和自适应注意力引导神经块(AGN)。
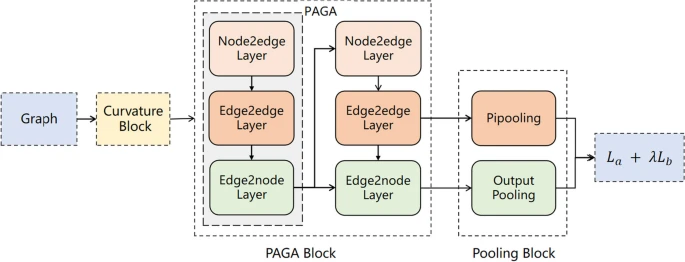
曲率块分配边缘属性以包含多尺度曲率,AGN 受到 SIGN 的启发,由两部分组成。第一部分称为极坐标自适应图注意模块 (PAGA),它使用自适应图注意机制通过结合距离、角度和曲率信息来对蛋白质-配体复合物的 3D 空间结构进行建模。自适应注意机制解决了蛋白质-配体复合图中的异质性。第二部分是池化模块,其中描述并包括用于利用长程相互作用的成对交互池化 (PiPool) 和用于预测蛋白质-配体结合亲和力的输出池化层。
并且,由于节点属性对图结构的依赖不同,研究人员在 PAGA 的edge2edge层中使用向量注意力,这使得模型能够针对节点中的不同属性学习不同的注意力权重。此外,由于节点属性对图结构的依赖不同,该团队在 PAGA 的 edge2edge 层中使用向量注意力,这使得模型能够针对节点中的不同属性学习不同的注意力权重。
研究人员在标准 PDBbind-v2016 数据集上训练模型,其实验结果在 RMSE 上优于 SIGN 7.5%,在 MAE 上优于 SIGN 9.4%,这证实了所提出的 CurvAGN 模型在改善蛋白质-配体结合亲和力预测方面是有效的。对于蛋白质-配体结合亲和力预测,预测的准确性对于药物的设计和开发、理解蛋白质功能和相互作用机制等非常重要。因此,即使 RMSE 的提升很小,该方法也可以提高预测的准确性并提供更可靠和有用的结果。
研究人员认为,有必要进行进一步的探索来解决该模型可能无法提高所有蛋白质-配体复合物预测准确性的问题。此外,该团队的目标是将复合体的整体几何信息(例如拓扑信息)纳入网络结构中。最后,研究人员希望该模型应用到生物学的其他领域,例如 miRNA 疾病关联预测和药物重新定位。
论文链接:https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-023-05503-w

